Número Atual: Julho-Setembro 2019 - Volume 3 - Número 3
- Imprimir
- Indicar
- Estatísticas
- (0)
Comentários - Como Citar
- Download da Citação
- Artigos Relacionados
-
Outros dos
Autores
Artigo Original
Associação do polimorfismo de IL-13 (+1398A/G) com a asma e a ancestralidade genômica
Association of IL-13 (+1398A/G) gene polymorphisms with asthma and genomic ancestry
Sonia M. T. Reis1; Luciano G. da S. Gomes1; Ohana L. S. de Oliveira1; Lauro J. Marin2; Marco A. Costa1,3; Sandra R. Gadelha3; Sandra M. B. Sousa4
DOI: 10.5935/2526-5393.20190039
1. Universidade Estadual de Santa Cruz, Departamento de Ciências Biológicas, Programa de Pós-graduação em Genética e Biologia Molecular - Ilhéus, BA, Brasil
2. Universidade Estadual de Santa Cruz, Departamento de Ciências da Saúde - Ilhéus, BA, Brasil
3. Universidade Estadual de Santa Cruz, Departamento de Ciências Biológicas - Ilhéus, BA, Brasil
4. Universidade Estadual do Sudoeste da Bahia, Departamento de Ciências Naturais - Vitória da Conquista, BA, Brasil
Endereço para correspondência:
Sandra M. B. Sousa
E-mail: marasbs@yahoo.com.br
Não foram declarados conflitos de interesse associados à publicação deste artigo.
Submetido em: 18/03/2019
Aceito em: 03/06/2019
RESUMO
INTRODUÇÃO: A asma é uma doença complexa, resultante da interação entre fatores genéticos e ambientais. A expressão aumentada de genes relacionados à inflamação define as alterações celulares e estruturais do aparelho respiratório, enquanto o meio ambiente modula os diferentes fenótipos asmáticos. Os produtos dessas células envolvidos na inflamação incluem citocinas, como a interleucina13 (IL-13), que está relacionada com a síntese direta de IgE, imunoglobulina essencial na patogênese da asma. Há divergências entre a prevalência da asma e o grupo étnico estudado, desta forma, o uso de Marcadores Informativos de Ancestralidade (AIM - Ancestry Informative Markers) possibilita a caracterização da ancestralidade genômica de diferentes populações.
OBJETIVOS: Verificar a associação entre polimorfismos do gene IL-13R com a ancestralidade genômica e a asma em uma população no sul da Bahia.
MÉTODOS: Foram genotipadas 320 amostras, sendo 114 casos, e 206 controles, utilizando o método de PCR e PCR/RFLP em sete AIMs (Sb19.3, APO, AT3, RB2300, LPL, CKMM e PV92) que apresentam elevado diferencial de frequência alélica entre africanos, ameríndios e europeu, e um polimorfismo no receptor de IL-13 (IL-13RA1).
RESULTADOS: Os resultados desse estudo mostraram que a maior contribuição foi ameríndia, tanto para os casos (37,42%), como para os controles (50,52%), demonstrando que há diferenças nas contribuições étnicas das amostras da região estudada. O polimorfismo no receptor de IL-13 (IL13RA1) apresentou associação significativa com rinite e história familiar.
CONCLUSÕES: A heterogeneidade da composição étnica das amostras pode ter influenciado na não associação das duas variáveis: níveis de IgE sérico e histórico familiar, e a presença do polimorfismo no receptor da IL-13RA1, e aponta a necessidade de realização do controle genômico.
Descritores: AIM, marcadores moleculares, IL13, doença inflamatória crônica, vias aéreas inferiores.
Introdução
A asma é uma doença inflamatória crônica que provoca hiper-responsividade das vias aéreas inferiores, limitando o fluxo aéreo. Sua origem não é totalmente elucidada, e envolve mecanismos genéticos, bem como fatores ambientais no desencadear da patogênese1. No Brasil, a asma é um relevante problema de saúde pública, e aparece entre as doenças mais frequentemente relacionadas com atendimentos em emergências e hospitalizações2. A prevalência de asma na população brasileira atinge aproximadamente 10%, sendo este um valor inferido, considerando o número de estudos epidemiológicos em âmbito nacional ser incipiente3. A frequência de asma ativa, grave e diagnosticada entre escolares e adolescentes em diversas cidades brasileiras aponta que na Região Nordeste do Brasil, os níveis de prevalência da asma variam de 14,8 a 30,5%2.
A asma demonstra ser uma condição complexa, e a diversidade de mecanismos genéticos envolvidos na determinação da doença ainda não foi totalmente elucidada. Diversos polimorfismos em genes candidados já foram identificados, dentre eles IL4, IL13, STAT6, GSDML, ADRB2 e ADAM33, porém poucos estudos trazem a associação destes com a ancestralidade genômica4,5.
O polimorfimo no gene do receptor da IL-13RA1 (+1398A/G) está diretamente relacionado com o desenvolvimento da asma, e é caracterizado pela substituição de uma adenina (A) por uma guanina (G), tendo como genótipos possíveis AA, AG e GG. O IL-13RA1 codifica uma proteína composta de 427 aminoácidos que exerce funções de sinalização, tanto para a IL-13, quanto alternativamente para a IL-46. A susceptibilidade genética colabora para o risco da asma, porém pouca associação genética foi encontrada em populações não europeias e sul-americanas7-12.
A susceptibilidade a asma ou a qualquer aspecto relacionado a esta patologia e a sua associação com a ancestralidade dos indivíduos é essencial na determinação de sua prevalência em grupos étnicos específicos. Para tanto, pode se utilizar marcadores informativos de ancestralidade (AIMs) com um valor de diferencial de frequência mínimo (30%) entre ao menos duas populações definidas geográfica e/ou etnicamente10,13-15.
A população brasileira é diversificada geneticamente, resultado da miscigenação entre ameríndios, afrodescendentes e euro-descendentes2. Considerando os mais de 500 anos de mistura genética, utilizar apenas a cor da pele ou a autodenominação como meio de classificação étnico-racial não abrange todas as nuances dessa mistura, e sua associação com doenças2.
Os resultados de associação da asma e a ancestralidade genômica em diversos estudos utilizando os AIMs podem ser altamente divergentes4,16,17, sendo que a discrepância entre os estudos pode ocorrer devido à composição genética de cada grupo ancestral, onde cada grupo étnico envolvido pode apresentar frequências alélicas diferentes, tornando certas populações mais susceptíveis a determinadas patologias4,8,18-21.
O presente trabalho teve por objetivo verificar a associação e a frequência do polimorfismo genético de IL-13RA1 (+1398A/G) com a ancestralidade genômica e a asma, em uma população do sul do estado da Bahia, Brasil.
Métodos
Declaração ética
O estudo foi do tipo caso-controle, aprovado pelo Comitê de Ética da Universidade Estadual de Santa Cruz - UESC (Protocolo: 453/2011). Os dados foram obtidos com a aplicação de um questionário e através da coleta de sangue periférico.
Grupo amostral
O grupo caso foi formado por pessoas asmáticas, e o grupo controle composto por pessoas que não sofrem dessa condição. O recrutamento dos casos ocorreu em clínicas particulares de pneumologia na cidade de Itabuna/Bahia/Brasil, sendo utilizados como critério de inclusão os pacientes que apresentavam asma diagnosticada por esses especialistas. Houve o cuidado da distinção entre pacientes asmáticos e pacientes com DPOC (Doença Pulmonar Obstrutiva Crônica), sendo este último o primeiro fator de exclusão para esse grupo. O diagnóstico foi baseado na anamnese do paciente e condições clínicas, além da função pulmonar (espirometria), entretanto, não foram coletados dados deste último para este estudo. Destacamos que neste trabalho não houve subclassificação dos pacientes asmáticos quanto seus fenótipos e/ou endofenótipos. Entretanto, podemos afirmar que 60% dos pacientes apresentaram IgE total acima de 160 UI/mL, sugerindo dentre estes asma decorrente da rota imunológica Th2. Todos os pacientes, tanto casos como controles, foram inqueridos sobre serem alérgicos, com o uso de questionário. Os casos considerados positivos eram contabilizados condicionalmente após confirmação clínica. A maior parte desses pacientes alérgicos declarou ter feito testes cutâneos de sensibilidade a alérgenos (prick test).
Os indivíduos controles foram recrutados em laboratório de análises clínicas também do município de Itabuna/Bahia/Brasil, sendo selecionados pelos pesquisadores por estarem realizando exames de rotina ou exames admissionais, desde que não apresentassem asma ou relato de qualquer problema respiratório.
Como critério de exclusão, foi adotado o tabagismo, excluindo-se assim todo e qualquer paciente/ controle que acusou fumar mais de 100 cigarros em sua vida e/ou alegaram estar atualmente fumando, ainda que ocasionalmente.
Marcadores Informativos de Ancestralidade (AIMs)
Foram analisados sete marcadores que se enquadram na definição de AIMs, cinco (RB2300, LPL, AT3-I/D, Sb19.3 e APO) com valores de δ > 40% entre Africanos e Europeus ou Ameríndios14,22, e dois (PV92 e CKMM) com δ > 40% entre Ameríndios e Africanos ou Europeus15. Para os loci analisados no presente estudo, a denominação de alelo *1 (ex., APO *1) foi dada ao fragmento correspondente à banda de maior peso molecular observada no gel de agarose, devido à presença de uma inserção (para os loci AT3-I/D, APO, PV92 e Sb19.3) ou à ausência de um sítio de restrição (para os loci, RB2300, CKMM e LPL)22.
Foram genotipadas 320 amostras, sendo 114 casos e 206 controles, utilizando: sete pares de primers para os sete marcadores, 4 μL do DNA genômico, previamente extraído, e 21 μL do MIX contendo os demais reagentes (água, buffer, dNTP, primers, MgCl², Taq polimerase) perfazendo um volume total de 25 μL na Reação em Cadeia Polimerase (PCR). Para cada análise foi usado um controle negativo contendo água no lugar do DNA genômico, segundo as condições descritas por Parra e cols. (1998), com pequenas modificações.
Os SNPs (LPL, CKMM, RB2300) foram analisados pela técnica da PCR-RFLP. O indivíduo foi denominado homozigoto para a presença do sítio de restrição da enzima (genótipo *2/*2), heterozigoto (genótipo *1/*2), ou homozigoto para a ausência do sítio de restrição (genótipo *1/*1). O controle utilizado foi o DNA amplificado de um indivíduo de genótipo conhecido. Os produtos amplificados foram separados em géis de agarose (2 e 3%), os quais foram visualizados em fotodocumentador com o uso de luz UV para análise do perfil eletroforético.
Determinação do polimorfismo IL-13RA1 (+1398A/G)
Foram utilizados os iniciadores: Forward - 5'- TCA GTG ATG GAG ATA ATT TA-3' e Reverse -5'-TGA GCT GCC TGT TTA TAA AT-3', que amplificam um fragmento de 130 pb. A reação de PCR com volume total de 25 μL (21 μL dos demais reagentes + 4 μL de DNA) seguiu com 35 ciclos em termociclador. A detecção do SNP no gene IL-13RA1, por meio da análise dos fragmentos de restrição, foi realizada pela mistura de 4 μL do produto de PCR e 6 μL da mistura de solução para RFLP. Esta mistura continha tampão e a endonuclease de restrição, MseI 5U, incubada a 37º C por 4 horas. Os fragmentos de 85 e 45 pares de base foram visualizados em gel de agarose 1,5%23.
Análise dos dados
Utilizou-se o programa Genepop24 para calcular as frequências alélicas e os desvios do equilíbrio de Hardy-Weinberg para os AIMs. A estimativa de mistura genética foi calculada com o programa Admix 3 e 2. As análises das frequências alélicas e genotípicas do IL-13RA1 e a associação entre acometimentos da asma e o polimorfismo no receptor de interleucina 13 (IL-13RA1) foram realizadas no programa SPSS 20.0 e Microsoft Excel®.
Resultados
Análise da ancestralidade genômica em amostras populacionais do sul da Bahia
Com o objetivo de analisar a ancestralidade genômica dos indivíduos asmáticos e não asmáticos na região sul da Bahia foram genotipados sete AIMs. Foram observados todos os alelos já identificados para os sete loci utilizados (APO, SB19.3, PV92, AT3, LPL, RB2300 e CKMM). A comparação das distribuições das frequências alélicas entre as duas amostras, mostrou que os alelos LPL*1, APO*1 e Sb19.3*1 foram mais frequentes nas amostras casos (asmáticos) quando comparadas com os controles (não asmáticos). Contrariamente, o alelo AT3*1 foi mais frequente entre os controles. Os alelos RB2300*1, CKMM*1 e PV92*1 também foram mais frequentes entre os controles, mas não foi observada diferença estatística significativa (Figura 1).
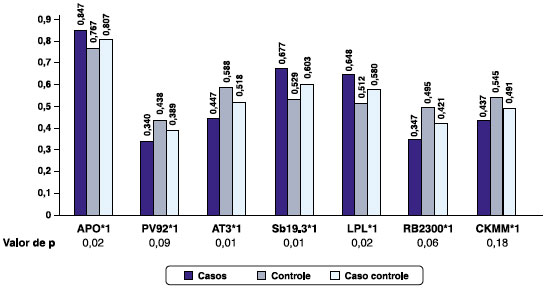
Figura 1 Frequências genotípicas e alélicas dos sete loci nas duas amostras populacionais analisadas
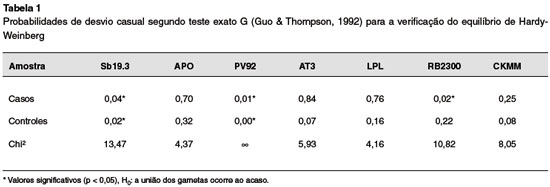
As frequências genotípicas observadas dos sete loci foram comparadas aos valores esperados segundo o equilíbrio de Hardy-Weinberg pelo teste exato G, onde se observou cinco desvios no geral, dois nos loci Sb19.3 e PV92 para casos e controles, e um no locus RB2300 para os casos (Tabela 1). Testes mais sensíveis foram utilizados para detectar se os desvios observados ocorreram pela ausência ou pelo excesso de heterozigotos. Os desvios nos loci Sb19.3 e PV92 podem ser explicados pela falta de heterozigotos, enquanto que o locus RB2300 mostrou desvios explicados pelo excesso de heterozigotos.
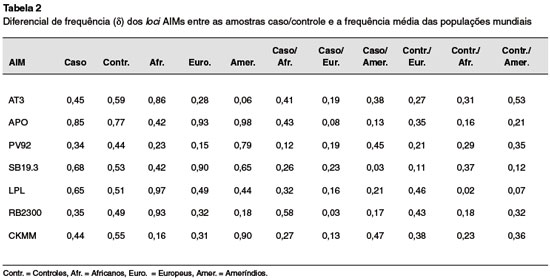
Os valores de diferencial de frequência (δ) entre as amostras analisadas e os grupos mundiais originais estão apresentados na Tabela 2. Os valores dos sete loci analisados foram eficazes na separação entre os grupos, pois os maiores valores de δ pertenceram aos grupos étnicos propostos para a diferenciação genética. Os loci AT3, RB2300 e CKMM apresentaram maior diferencial de frequências entre todos os loci analisados (Tabela 2).
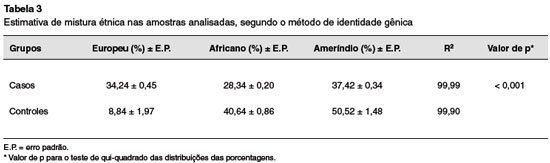
Os valores percentuais das estimativas de mistura étnica foram diferentes em cada um das amostras populacionais e foram consistentes com o modelo tri-híbrido, além de indicar uma maior contribuição ameríndia em todas as amostras: casos (37,42%) e os controles (50,52%) (Tabela 3). Além disso, observouse que no grupo caso há maior homogeneidade nas contribuições étnicas do que no grupo controle.
Polimorfismo no gene IL-13 e sua associação com a asma
O polimorfismo IL-13RA1 em estudo (+1398A/G) é caracterizado pela substituição de uma adenina por uma guanina, tendo como genótipos possíveis AA, AG e GG. Como não foram identificados em nossa amostragem indíviduos heterozigotos, as frequências alélicas e genotípicas apresentaram os mesmos valores: genótipo AA/alelo A (62%) e genótipo GG/alelo G (38%) para os genótipos selvagem e mutante, respectivamente.
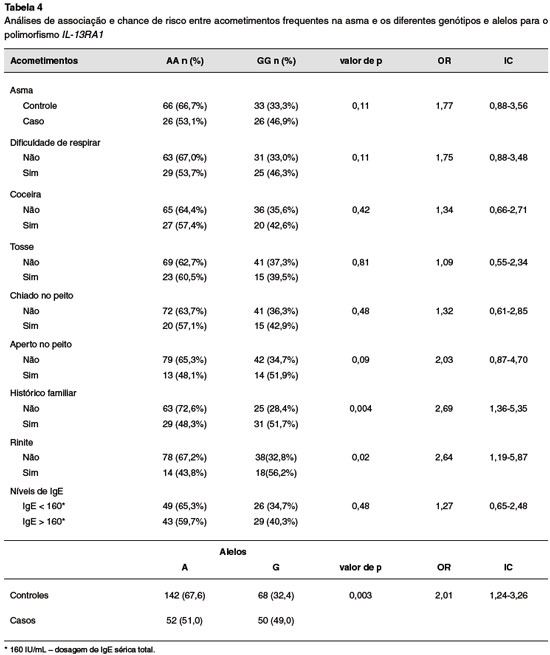
Nas análises de associação das variáveis analisadas com o polimorfismo da IL-13RA1 foi possível observar associação significativa com a história familiar, mais não com os níveis de IgE, segundo o valor de corte pré-estabelecido de 160 IU/mL. Quanto aos diversos acometimentos conjuntos com a asma, foi possível verificar que secreção nasal e espirros constantes demonstraram correlações estatísticas com esse polimorfismo (respectivamente, p = 0,004, OR = 2,88, IC = 1,39-5,99; p = 0,04, OR = 2,09, IC = 1,05-4,16). Foi possível resumir essas duas variáveis em histórico de rinite, cujos resultados de sua associação com o polimorfismo encontram-se na Tabela 4. Apesar de não apresentar associação significativa entre os grupos caso e controle e o genótipo, foi observada associação significativa entre esses grupos e os alelos, tendo uma chance de risco 2 vezes mais elevada para quem apresenta o alelo *G (Tabela 4).
Discussão
O processo de mistura étnica da população brasileira tem sua característica própria devido à origem das populações que se estabeleceram no país. Os AIMs, por sua vez, são importantes marcadores biológicos, pois fornecem resultados mais precisos sobre o perfil genético ancestral de uma determinada população. Desse modo, as frequências alélicas das amostras em relação às populações mundiais podem explicar graus diferentes de miscigenação do que se é esperado meramente pela cor da pele, por exemplo13,22.
Inicialmente a não aderência ao equilíbrio de Hardy-Weinberg dos loci Sb19.3, PV92 e RB2300 pode ser devido ao pequeno tamanho amostral, e principalmente por possuírem uma característica específica: serem asmáticos; portanto, uma amostragem não aleatória.
O estudo de marcadores de ancestralidade genômica em amostras aleatórias representativas da população de Salvador, capital do estado da Bahia, coletadas em diversas regiões da cidade, evidenciou que, apesar de não preencher todos os critérios teóricos, a população de Salvador estava em equilíbrio de Hardy-Weinberg25. Outra possível explicação para a amostra não estar em equilíbrio, é a existência de "reprodução preferencial" já demonstrada em estudos realizados no estado da Bahia, devido à tendência dos indivíduos de se casarem dentro de grupos fenotipicamente similares26.
Quanto às estimativas de mistura étnica obtidas utilizando o método da identidade gênica27, o seu coeficiente de identidade gênica estima a probabilidade de dois genes escolhidos ao acaso (em uma ou mais populações) serem idênticos por natureza. Dessa análise, revelou-se maior contribuição ameríndia nesta amostra populacional do sul da Bahia, mais precisamente de Itabuna/BA/Brasil, tanto nos casos quanto nos controles que compõem o estudo.
Desde o início da colonização e povoamento da região, a população indígena sofreu às uniões preferenciais entre homens africanos e europeus e mulheres ameríndias25. Entretanto, as porcentagens de contribuição étnica são significativamente desiguais entre casos e controles. Intrigantemente, o grupo caso apresentou uma herança homogênea da tríade étnica que origina a população miscigenada dessa região. Estudos anteriores, com pacientes asmáticos e AIMs, concluíram que a etnia negra confere maior suscetibilidade à asma dentre os indivíduos do grupo caso4,10,11. Em contrapartida, é possível que indivíduos da nossa amostra populacional, com maior ancestralidade genômica europeia, estejam ligados à maior taxa percentual de indivíduos asmáticos aqui nesta região da Bahia.
Essas contraposições realçam a complexidade da asma. Não obstante, ainda é possível que a ancestralidade genômica não confira vulnerabilidade para a asma nessas amostras do sul do estado da Bahia, mas sim a presença ou ausência de outras diversas variáveis. A asma é uma patologia complexa, e o estudo da relação entre a doença e o histórico familiar e níveis de IgE sérico são pontos importantes para subclassificar a asma (seus fenótipos e endofenótipos) e fechar o diagnóstico adequado ao quadro do paciente, e, assim, atribuir o tratamento especializado29. A análise do polimorfismo no receptor da IL-13RA1 e a susceptibilidade para asma nesta amostra não mostrou associação entre essas duas variáveis, provavelmente o tamanho amostral tenha influenciado esse resultado.
O fator histórico familiar de asma mostrou ser um forte indicador de suscetibilidade na presença do polimorfismo no IL-13RA1. Os fatores genéticos, por exemplo, são tão importantes no quadro asmático que a herdabilidade pode chegar próximo a 80%28. Deste modo, tais fatores são generosamente discutidos no ciclo da asma, pois ter uma história familiar de asma, em teoria, aumenta a probabilidade do desenvolvimento da doença.
Os sintomas frequentes relacionados à asma, como o quadro de rinite, apresentaram associação significativa com o polimorfismo (IL-13RA1) nesse estudo, tais sintomas estão fortemente presentes em indivíduos acometidos pela doença. De fato, muitos estudos mostram que a rinite alérgica e a asma são doenças inter-relacionadas, sendo que a rinite ocorre em 75 a 90% dos indivíduos adultos com asma alérgica28.
É importante salientar que o presente estudo apresenta certas limitações. Primeiro, o tamanho amostral limitado para análises de populações miscigenadas e a condição complexa da asma, pode levar à perda de acurácia na associação investigada. Segundo, a carência de precisa caracterização dos pacientes asmáticos, visto que não se levou em conta os diferentes fenótipos da doença. Entretanto, acreditamos que os achados aqui expostos podem gerar novos insights para futuros estudos envolvendo asma e as diferentes etnias que compõem a população baiana.
Neste estudo, utilizando AIMs, foi observada a alta miscigenação em amostras da região do sul da Bahia. Essas proporções refletem contribuições diferentes das populações ancestrais no processo de miscigenação. Diferentemente do esperado pela história de colonização da Bahia, que recebeu grande contingente de africanos, como mão de obra escrava para os latifúndios, esperava-se uma maior contribuição africana, no entanto uma maior ancestralidade ameríndia foi observada nas amostras, provavelmente o processo de interiorização dos índios do litoral para o interior do estado, devido às invasões marítimas estrangeiras, tenha contribuído fortemente para esses achados. Desta forma, a ancestralidade genômica é uma variável a ser analisada em estudos de doenças multifatorial e complexa como a asma e em amostras latino-americanas para realização do controle genômico, tendo em vista que em outras populações de etnias africanas a asma está mais prevalente, embora tenha divergências4,16,17.
Agradecimentos
Agradecemos ao Programa de Pós-graduação em Genética e Biologia Molecular da Universidade Estadual de Santa Cruz; à CAPES, pela bolsa de estudo; ao laboratório LAP, representado pelo Sr. Ari Paranhos; ao Laboratório LIDI, representado pelo Sr. Neto; ao Laboratório Álvaro, representado pelo Sr. Gustavo; aos pneumologistas Dra. Rosângela, Dr. Gustavo, Dr. Silvane, Dr. Laurindo e Dr. Clóvis; e também ao CNPq e à FAPESB pelo apoio financeiro.
Referências
1. Bont L. Bronquiolite e asma: o próximo passo. J Pediatr (Rio J.). 2017;93(3):209-10.
2. Solé D, Camelo-Nunes IC, Wandalsen GF, Mallozi MC. A asma na criança e no adolescente brasileiro: contribuição do International Study of Asthma and Allergies in Childhood (ISAAC). Rev Paul Pediatr. 2014;32(1):114-25.
3. Stelmach R, Cruz AA. O paradoxo da asma: negligência, fardo e big data. J Bras Pneumol. 2017;43(3):159-60.
4. Vergara C, Caraballo L, Mercado D, Jimenez S, Rojas W, Rafaels N, et al. African ancestry is associated with risk of asthma and high total serum IgE in a population from the Caribbean Coast of Colombia. Hum Genet. 2009;125(5-6):565-79.
5. Berenger AG, Fernandes AT, Oliveira S, Rodrigues M, Ornelas P, Romeira D, et al. Genetic polymorphisms and asthma: findings from a case-control study in the Madeira island population. BMC Biol Res. 2014;47:40.
6. Ding G, Ruouxu J, Bao Y. Risk and Protective Factors for the Development of Childhood Asthma. Paed Resp Rev. 2015;16(2):133-9.
7. Seyfizadeh N, Seyfizadeh N, Gharibi T, Babaloo Z. Interleukin-13 as an important cytokine: A review on its roles in some human diseases. Acta Microb Immuno Hungar. 2015;62(4):341-78.
8. Choudhry S, Coyle NE, Tang H, Salari K, Lind D, Clark SL, et al. Population stratification confounds genetic association studies among Latinos. Hum Genet. 2006,118:652.
9. Mathias RA, Grant AV, Rafaels N, Hand T, Gao L, Vergara C, et al. A genome-wide association study on African-ancestry populations for asthma. J. Allergy ClinImmunol. 2010;125:336-46.
10. Barnes KC, Grant AV, Hansel NN, Gao P, Dunston GM. African Americans with asthma: genetic insights. Proc Am Thorac Soc. 2007;4:58-68.
11. Baye TM, ButschKovacic M, Biagini Myers JM, Martin LJ, Lindsey M, Patterson TL, et al. Differences in Candidate Gene Association between European Ancestry and African American Asthmatic Children. PLOS ONE. 2011;6(2):e16522.
12. Hussein YM, Shalaby SM, Mohamed RH, Hassan TH. Association between genes encoding components of the IL-10/IL-0 receptor pathway and asthma in children. Ann Allergy Asthma Immunol. 2011;106(6):474-80.
13. Parra EJ, Kittles RA, Argyropoulos G, Pfaff CL, Hiester K, Bonilla C, et al. Ancestral proportions and admixture dynamics in geographically defined African Americans living in South Carolina. Am J PhysAnthropol. 2001;114(1):18-29.
14. Shriver MD, Parra EJ, Dios S, Bonilla C, Norton H, Jovel C, et al. Skin pigmentation, biogeographical ancestry and admixture mapping. Hum Genet. 2003;112(4):387-99.
15. Bonilla C, Parra EJ, Pfaff CL, Dios S, Marshal JA, Hamman RF, et al. Admixture in the Hispanics of the San Luis Valley, Colorado, and its implications for Complex trait gene mapping. Ann Hum Genet. 2004;68:139-53.
16. Mersha TB, Abebe T. Self-reported race/ethnicity in the age of genomic research: its potential impact on understanding health disparities. BMC Hum Genomics. 2015,9:1.
17. Ahmedani BK, Peterson EL, Wells KE, Rand CS, Williams LK. Asthma medication adherence: the role of God and other health locus of control factors. Ann Allergy Asthma Immunol. 2013;110(2):75-9.
18. Salam MT, Avoundjian T, Knight WM, Gilliland FD. Genetic Ancestry and Asthma and Rhinitis Occurrence in Hispanic Children:Findings from the Southern California Children's Health Study. PLOS ONE. 2015;10(9):e0139397.
19. Salari K, Shweta C, Tang H, Naqvi M, Lind D, Avila PC, et al. Genetic admixture and asthma-related phenotypes in Mexican American and Puerto Rican asthmatics. Gen Epidemiol. 2005;29(1):76-86.
20. Haller G, Torgerson DG, Ober C, Thompsom EE. Sequencing the IL4 locus in African Americans implicates rare noncoding variants in asthma susceptibility. J Allergy Clin Immunol. 2009;124(6):1204-9.
21. Jain S, Fajt ML, Wenzel S. Does level of allergen sensitization impact asthma related outcomes differently in adult and pediatric asthma patients? J Allergy Clinic Immunol. 2018;141(2):AB220.
22. Parra EJ, Marcini A, Akey J, Martinson J, Batzer MA, Cooper R, et al. EstimatingAfrican American admixture proportions by use of population-specific alleles. Am J Hum Genet. 1998;63(6):1839-51.
23. Kim H, Lee H-C, Lee S-Y, Jung J, Jin H-S, Kim J-H, et al. Genegene interaction between IL-13 and IL-13Ra1 is associated with total IgE in Korean children with atopic asthma. J Hum Genet. 2006;51:1055-62.
24. Raymond M, Rousset F. GENEPOP (version 1.2): population genetics software forexact tests and ecumenicism. J Hered. 1995;86:248-9.
25. Machado TMB, Bomfim TF, Acosta AX, Galvão-Castro B, Abé-Sandes K. Estimativa de mistura genética em uma amostra da população de Salvador-BA-Brasil. FIOCRUZ-BA, 2007.
26. Abe-Sandes K, Bomfim TF, Abe-Sandes C, Acosta AX, Alves CRB. Ancestralidade genômica, nível socioeconômico e vulnerabilidade ao HIV/AIDS na Bahia, Brasil. Saúde Soc São Paulo. 2010;19(supl.2):75-84.
27. Chakraborty R, Kamboh MI, Nwankwo M, Ferrell RE. Caucasian genes in American blacks: new data. Am J HumGenet. 1985;50:145-55.
28. Koppelman GH, Los H, Postma DS. Genetic and environment in asthma: the answer of twin studies. Eur Respiratory J. 1999;13(1):2-4.
29. Carr TF, Zeki AA, Kraft M. Eosinophilic and noneosinophilic asthma. Am J Respir Crit Care Medice. 2018;197(1):22-37.